KI designt neue Proteine, die in Labortests Bakterien töten
Das Deep-Learning-Sprachmodell ProGen kann Aminosäure-Sequenzen für Eiweiße mit gewünschter Funktion entwerfen. Dafür muss es keine Proteinfaltregeln kennen.
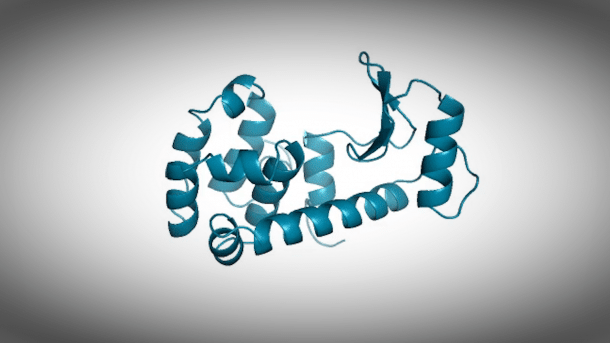
Struktur eines funktionalen künstlichen Proteins, das die KI von ProGen entworfen hat. Der Vertreter aus der Familie der Lysozyme ist zu 69 Prozent mit einem natürlichen Protein identisch.
(Bild: Salesforce AI Research)
- Veronika Szentpetery-Kessler
Eine künstliche Intelligenz (KI) namens ProGen hat neue antibakterielle Proteine entworfen, die in Labortests Erreger erfolgreich abgetötet haben. Die designten Eiweiße sind der Enzym-Gruppe der Lysozyme nachempfunden. Die natürlichen Vorbildern zerlöchern die Zellwand von Bakterien und kommen etwa in Tränen, Speichel und der Muttermilch vor.
Videos by heise
Die von Salesforce AI Research in den USA entworfenen Enzymvarianten gibt es in der Natur so nicht. Sie haben aber große strukturelle Ähnlichkeiten mit ihren biologischen Vorbildern, schreiben die Salesforce AI Research-Entwickler gemeinsam mit Kooperationspartnern vom kalifornischen Biotech-Unternehmen Profluent und von der University of California in San Francisco im Fachjournal "Nature Biotechnology".
Von einer Million neu generierten Lysozymen suchten sie 100 besonders naturähnliche Aminosäure-Sequenzen zur Synthese aus und untersuchten 90 von ihnen weiter. 73 Prozent der synthetischen Proteine (66 von 90) waren gegenüber einer Test-Zellwand antibakteriell wirksam, verglichen mit 59 Prozent der in der Studie verwendeten natürlichen Proteine (53 von 90). Zwei besonders wirksame Enzym-Kandidaten brachten auch lebende Escherichia coli-Bakterien um.
Die Faltkunst der Proteine
Proteine wie Enzyme bestehen aus Kombinationen von insgesamt 20 verschiedenen Aminosäuren, die zu Hunderten perlschnurartig verknüpft sind. Ihre Reihenfolge legt fest, wie sich die Kette dreidimensional faltet, da die Aminosäuren verschiedene Seitengruppen und Ladungen tragen. Die bestimmen, welche Aminosäuren sich gegenseitig anziehen oder auch abstoßen. Die finale Form entscheidet anschließend über die Funktion und Wirksamkeit der Proteine.
Um die neuen Lysozyme zu entwerfen, musste das Deep-Learning-Sprachmodell von Salesforce allerdings nicht wissen, welche physikalisch-chemischen Eigenschaften Aminosäuren haben und wie sie miteinander interagieren. Stattdessen arbeitet es laut Teamleiter Nikhil Naik ähnlich wie Texte generierende KIs und betrachtet Proteine wie Artikel.
(Bild: Salesforce AI Research)
Ein Sprachmodell für Artikel würde man mit Texten über Themen wie Dichtkunst, Politik und Sport trainieren, sagt Naik. "Wenn man es also beauftragen würde, einen komplett neuen Sporttext zu schreiben, würde das Ergebnis sehr wahrscheinlich von Sport handeln", sagt Naik. Den Unterschied zwischen einem Sport- und einem Politikartikel mache das jeweils typische Vokabular aus.
Sprachmodell auf Aminosäuren trainieren
Genauso sei es bei ProGen. "Wenn es etwas Neues generiert, wird das die gleichen Eigenschaften haben wie die Enzymfamilien, an denen man interessiert ist und die man vorgibt“, erklärt Naik. Sein Team hatte das Sprachmodell mit knapp 56.000 Aminosäure-Sequenzen aus fünf unterschiedlichen Lysozym-Familien trainiert und ihm beigebracht, für welche Familie welche kurzen Aminosäure-Abfolgen an welchen Stellen der Kette typisch sind.
(Bild: Salesforce AI Research )
Davor hatten sie das Sprachmodell noch weiträumiger mit den Aminosäuren-Sequenzen von 280 Millionen Proteinen aus mehr als 19.000 Familien trainiert. Jede Sequenz erhielt zudem einordnende Etiketten (Tags) für Protein-Eigenschaften wie "antibakteriell" und die Art der Lysozym-Familie. Am Ende reichten der KI einige Tag-Vorgaben, um passende Aminosäureketten linear neu zu knüpfen. Dabei sagte ProGen anhand der wachsenden Abfolge jeweils die nächste wahrscheinlichste Aminosäure voraus.
Einsatz, um Medikamente zu entwickeln
Die ProGen-Methode könnte auch für die Entwicklung anderer Wirkstoffe nützlich sein. Sie wird zum Beispiel bereits zur Entwicklung von Medikamenten, die rheumatoide Arthritis und Multiple Sklerose bekämpfen sollen, eingesetzt, sagt AI Research.
Direktor Naik zufolge besteht der Hauptunterschied zu anderen, auf Proteine ausgerichteten KIs darin, dass ProGen Aminosäure-Sequenzen nicht nur lesen, sondern auch neu schreiben kann. Googles Deepmind-KI Alphafold hat letztes Jahr allein aus der Aminosäure-Abfolge die 3D-Struktur fast aller 200 Millionen Proteine vorhergesagt, die der Wissenschaft bekannt sind. Auch hier kannte die KI die physikalischen Gesetze der Proteinfaltung nicht. Experimentell bestätigt sind nur knapp 200.000 Proteinstrukturen. Dafür ist die aufwendige Synthese der Eiweiße und eine Strukturbestimmung per Kristallografie nötig.
Andere Teams nutzen sogenannte Diffusionsmodelle als generative KIs, um Entwürfe für teilweise sehr große Proteine mit gewünschten Formen und Eigenschaften zu generieren. Ihre Basis sind bekannte dreidimensionale Eiweißstrukturen. Das in Boston ansässige Start-up Generate Biomedicines hat im November des vergangenen Jahres ein Programm namens Chroma vorgestellt, das das Unternehmen als "DALL-E 2 der Biologie" bezeichnet. Anstatt Pixel zu manipulieren, arbeitet es mit zufälligen Aminosäureketten und setzt sie zu Strukturen mit gewünschten EIgenschaften zusammen. Dabei entfernt es unerwünschtes "Rauschen" aus den Daten, bis sozusagen ein klares Bild entsteht.
Parallel dazu hat ein Team an der University of Washington unter der Leitung des Biologen David Baker ein ähnliches Programm namens RoseTTAFold Diffusion entwickelt. Dieses nutzt mithilfe einer zweiten KI Informationen darüber, wie bestimmte Proteinstücke zusammenpassen, um die Endstruktur vorherzusagen.
(vsz)