Proteine statt Picasso: Von DALL-E inspirierte KI soll neue Medikamente erfinden
Zwei Forschergruppen planen leistungsstarke generative KI-Modelle, die neue Proteine entwerfen können, die in der Natur nicht vorkommen – etwa für die Medizin.
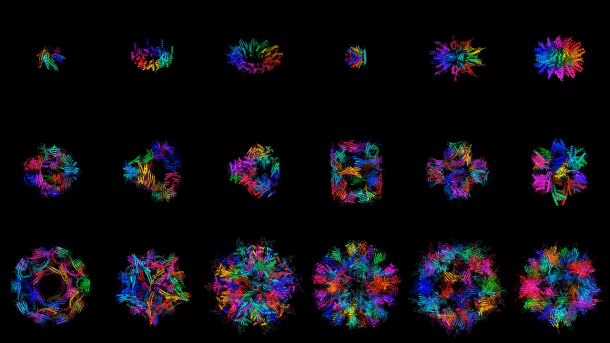
Proteinkomplexe.
(Bild: GENERATE BIOMEDICINES)
- Will Douglas Heaven
Text-zu-Bild-KI-Modelle wie DALL-E 2 von OpenAI werden immer besser. Sie sind darauf trainiert, Bilder von fast allem zu generieren, was man sich vorstellen kann. Das hat in der Kreativbranche – von der Mode bis zum Film – für Furore gesorgt, da die Technik auf Anfrage Bilder liefert, die so seltsam wie wunderbar sein können.
Die gleiche Technologie, die hinter diesen Algorithmen steht, scheint auch für Biotech-Labors interessant zu sein. Sie haben damit begonnen, die neue Art von generativer KI, die als sogenanntes Diffusionsmodell bekannt ist, zu verwenden, um ganz andere Dinge zu tun: neue, in der Natur nicht vorkommende Proteinarten, die für Medikamente verwendet werden können, zu entwickeln.
Videos by heise
Anfang Dezember zeigten zwei Labors unabhängig voneinander, wie das praktisch aussehen kann: Generate Biomedicines, ein in Boston ansässiges Start-up, stellte ein Programm namens Chroma vor, das das Unternehmen als das "DALL-E 2 der Biologie" bezeichnet. Parallel dazu hat ein Team an der University of Washington unter der Leitung des Biologen David Baker ein ähnliches Programm namens RoseTTAFold Diffusion entwickelt. In ihrem Preprint zeigen Baker und seine Kollegen, dass ihr Modell präzise Entwürfe für neuartige Proteine generieren kann, die sich dann im Labor zum Leben erwecken lassen. "Wir generieren Proteine, die keinerlei Ähnlichkeit mit vorhandenen Proteinen haben", sagt Brian Trippe, einer der Mitentwickler von RoseTTAFold.
Alles ganz neu und nur aus dem Rechner
Diese Protein-Generatoren können so gesteuert werden, dass sie Designs für Proteine mit bestimmten Eigenschaften – wie Form, Größe oder Funktion – erzeugen. Auf diese Weise ist es möglich, neue Proteine zu entwickeln, die bei Bedarf bestimmte Aufgaben erfüllen. Die Forscher hoffen, dass dies schließlich zur Entwicklung neuer und – deutlich – wirksamerer Medikamente führen wird. "Wir könnten in Minuten entdecken, wofür die Evolution Millionen von Jahren gebraucht hat", sagt Gevorg Grigoryan, CTO von Generate Biomedicines. "Das Bemerkenswerte an dieser Arbeit ist die Generierung von Proteinen nach vorab gewünschten Bedingungen", sagt Ava Amini, Biophysikerin bei Microsoft Research in Cambridge, Massachusetts, die die Arbeiten kennt.
Proteine sind die grundlegenden Bausteine der meisten Lebewesen. Bei Tieren helfen sie bei der Verdauung von Nahrung, kontrahieren Muskelen, detektieren Licht, steuern das Immunsystem und vieles mehr. Wenn Menschen krank werden, spielen Proteine eine zentrale Rolle. Sie sind daher bevorzugtes Ziel von Medikamenten. Und viele der neuesten Therapien basieren selbst auf Proteinen. "Die Natur verwendet Proteine für fast alles", sagt Grigoryan. "Die Möglichkeiten, die sich daraus für therapeutische Eingriffe ergeben, sind wirklich immens."
Unendlicher Pool an Eiweissen
Allerdings müssen die Entwickler von Medikamenten derzeit auf eine Liste von Inhaltsstoffen zurückgreifen, die aus natürlichen Proteinen bestehen. Das Ziel der Proteingenerierung ist es, diese Liste um einen nahezu unendlichen Pool von am Computer entworfenen Eiweissen zu erweitern. Solche Techniken sind nicht neu. Bisherige Ansätze waren jedoch langsam und nicht besonders gut geeignet, um große Proteine oder sogenannte Proteinkomplexe zu entwerfen – molekulare Maschinen, die aus mehreren miteinander verbundenen Proteinen bestehen. Genau diese Proteine sind oft entscheidend für die Behandlung von Krankheiten.
Die nun angekündigten KI-Systeme sind nicht die erste Nutzung von Diffusionsmodellen für die Proteinentwicklung. Eine Handvoll Studien, die Amini und andere in den letzten Monaten durchgeführt haben, hatten gezeigt, dass das Verfahren vielversprechend ist. Dennoch liegen bisher ausschließlich Proof-of-Concept-Prototypen vor. Chroma und RoseTTAFold Diffusion bauen auf diesen Arbeiten auf und sind die ersten "echten" Programme, die präzise Designs für eine Vielzahl von Proteinen erstellen können.